DIGITAL FUTURE
Introduzione al progetto
Come riportato da Masmec S.p.A.:
- Il progetto Digital Future introduce importanti risultati nella pratica chirurgica di natura mininvasiva, mettendo a disposizione del chirurgo tutto l’imaging intraoperatorio e preoperatorio in un unico ambiente di navigazione e consentendo così un approccio multimodale dell’area anatomica di interesse ed oggetto della procedura chirurgica.
- Le immagini provenienti dalle diverse sorgenti coesistono in uno stesso ambiente metrico e vengono sovrapposte mediante avanzati algoritmi di Image Processing che sono stati sviluppati nel corso del progetto. Oltre all’imaging intraoperatorio di natura dinamica (ecografica, endoscopia) e a quello diagnostico preoperatorio (TC, RM), sono state ricavate immagini derivate di tipo neurologico (Diffusion Tensor Imaging) di cui è stata riscontrata una notevole utilità chirurgica dalla comunità scientifica del settore.
- Infine, sono state implementate applicazioni in realtà aumentata mediante l’uso di visori olografici allo scopo di visualizzare una lesione tumorale o un’area anatomica al di là dell’ostacolo rappresentato dalla cute integra, a tutto vantaggio della mininvasività dell’atto chirurgico.
Contributo del Politecnico
Il Laboratorio di Informatica Industriale ha partecipato al progetto DIGITAL FUTURE come fornitore della Masmec S.p.A. per le due seguenti attività di ricerca e sviluppo:
- Metodologie di fusione di imaging diagnostico ed intraoperatorio real-time (ecografico ed endoscopico).
- Implementazione di algoritmi di fusione di imaging diagnostico ed intraoperatorio realtime (ecografico ed endoscopico).
Pubblicazioni
Il metodo principale per effettuare la segmentazione del parenchima e dei vasi è basato su Rete Neurale Convoluzionale, in particolare su una variante dell’architettura di V-Net.
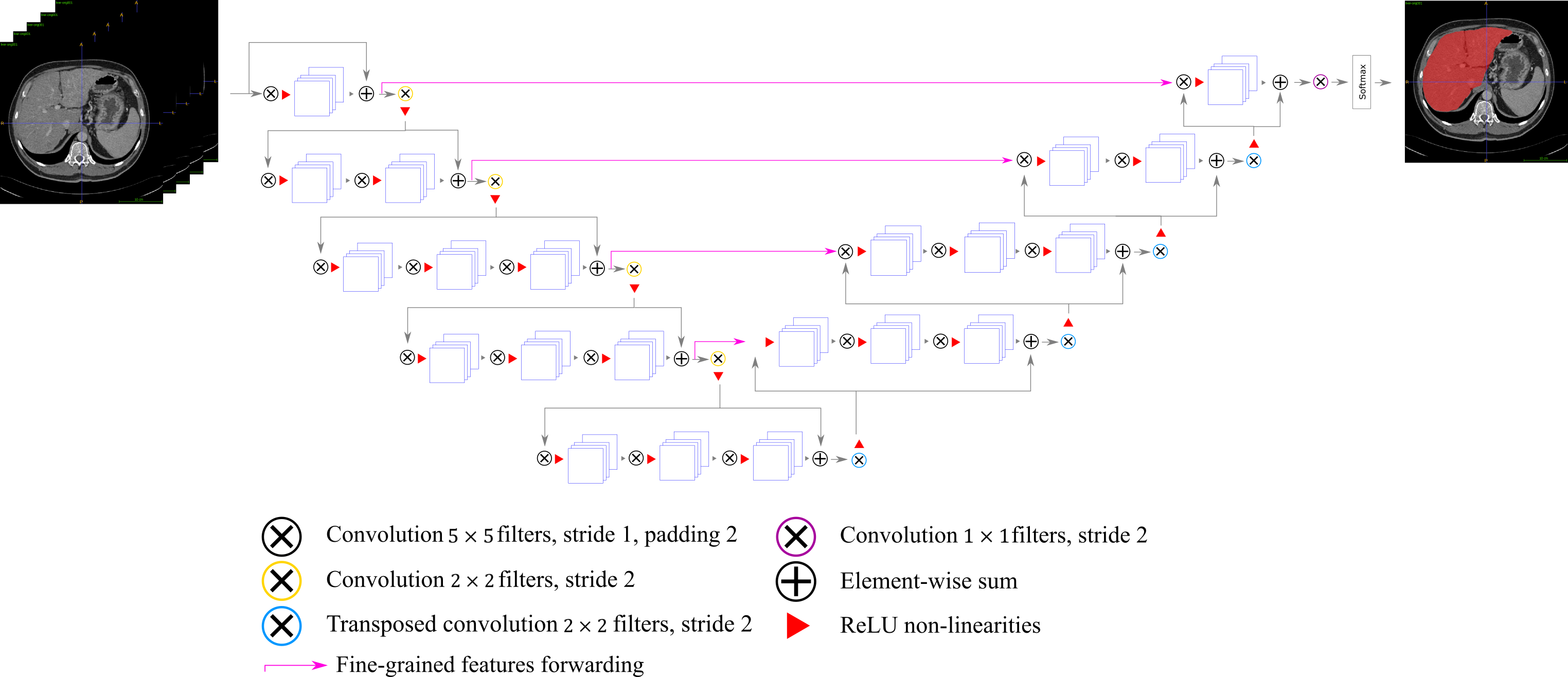
Nel post processing della fase di segmentazione del parenchima epatico sono stati utilizzati gli operatori morfologici per preservare soltanto la componente connessa più grande (basandosi sulla considerazione che il fegato è l’organo più grande presente nell’addome) e per chiudere l’immagine per evitare eventuali buchi.
Nel post processing della fase di segmentazione dei vasi epatici è stato effettuato un mascheramento per rilevare solo i vasi presenti all’interno del parenchima.
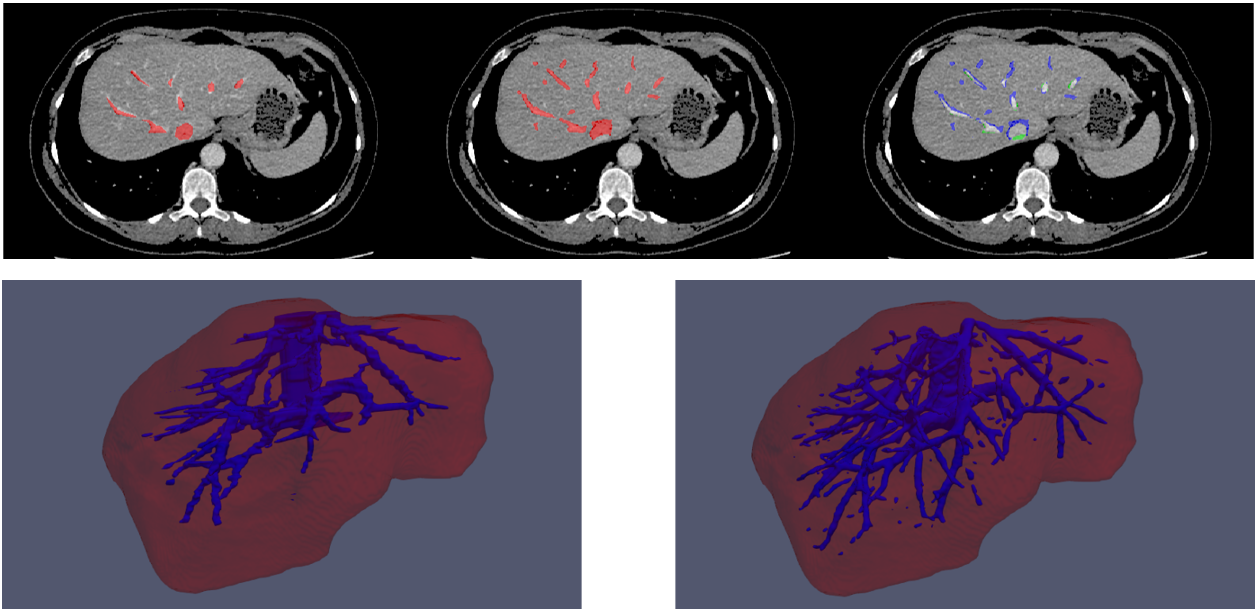
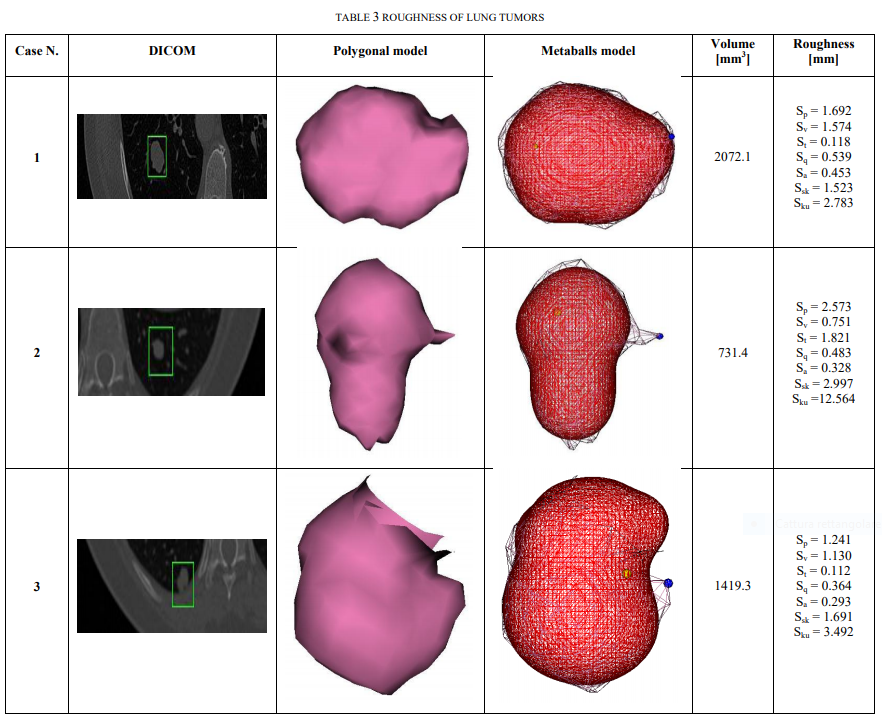
Il metodo è stato validato sia su dataset pubblici che locali, forniti dall’Unità di Radiologia d’Urgenza del Policlinico di Bari.
I risultati ottenuti sono stati molto promettenti, con un coefficiente di Dice maggiore del 96% per il modello di segmentazione del parenchima, e un accuratezza maggiore del 99% per il modello di segmentazione dei vasi, sui test set considerati.
A seguito del lavoro svolto, sono stati presentato due articoli scientifici alla 2020 International Conference on Intelligent Computing, October 2-5, 2020, Bari, Italy:
- Altini, N.; Prencipe, B.; Brunetti A.; Brunetti, G.; Triggiani, V.; Carnimeo, L.; Marino, F.; Guerriero, A.; Villani, L.; Scardapane, A.; Cascarano, G.D. A Tversky Loss-based Convolutional Neural Network for Liver Vessels Segmentation. In: Huang DS., Bevilacqua V., Hussain A. (eds) Intelligent Computing Theories and Application. ICIC 2020. Lecture Notes in Computer Science, vol 12463. Springer, Cham. https://doi.org/10.1007/978-3-030-60799-9_30
- Prencipe, B.; Altini, N.; Cascarano, G.D.; Guerriero, A.; Brunetti A. A Novel Approach based on Region Growing Algorithm for Liver and Spleen Segmentation from CT Scans. In: Huang DS., Bevilacqua V., Hussain A. (eds) Intelligent Computing Theories and Application. ICIC 2020. Lecture Notes in Computer Science, vol 12463. Springer, Cham. https://doi.org/10.1007/978-3-030-60799-9_35
Inoltre, è stata realizzata una survey a firma congiunta sulla prestigiosa rivista internazionale Neurocomputing:
- Altini, N.; Prencipe, B.; Cascarano, G.D.; Brunetti, A.; Brunetti, G.; Triggiani, V.; Carnimeo, L.; Marino, F.; Guerriero, A.; Villani, L.; Scardapane, A.; Bevilacqua, V. Liver, Kidney and Spleen Segmentation from CT scans and MRI with Deep Learning: A Survey. Neurocomputing, Volume 490, 2022, Pages 30-53, ISSN 0925-2312, doi:10.1016/j.neucom.2021.08.157.
Questi lavori seguono precedenti ricerche pubblicate in collaborazione la Masmec Spa volte all’individuazione, alla segmentazione e alla classificazione di lesioni tumorali da immagini TAC del distretto toracico.
- Bevilacqua, V., Ranieri, D., Nacci, G., Brunetti, G., Larizza, P., and Marino, F. (2014). An Innovative Soft Computing Framework to Measure and Classify Solid Pulmonary Tumors from CT Images. Journal of Bioinformatics and Biological Engineering, 2(1), 45-62. LINK